Přehled toho, co jsem zatím páchal v eRku
Nějak se mi stalo, že mám radši datovou analýzu pod Pythonem (Pandas/NumPy/Matplotlib), ale holt jsou ty užitečný věci pro proteomiku, transkriptomiku a genomiku napsaný v R. Bioconductor a tak. Stojíme na ramenou různě velkých obrů (občas když vidím složitější kód v R, jde na mě z velikosti těch obrů závrať).
Na svůj Github jsem umístil nějaké kódy v R, které můžou posloužit jako vodítko, nebo být přímo využité pro proteomické analýzy a metaanalýzy. Do značné míry jsou využitelné i pro expresní transkriptomická data.
https://github.com/hwllffrdd/proteomics
Zatím tu najdete:
- Comparison pro srovnání a korelaci dvou nezávislých kvantitativních proteomických datasetů
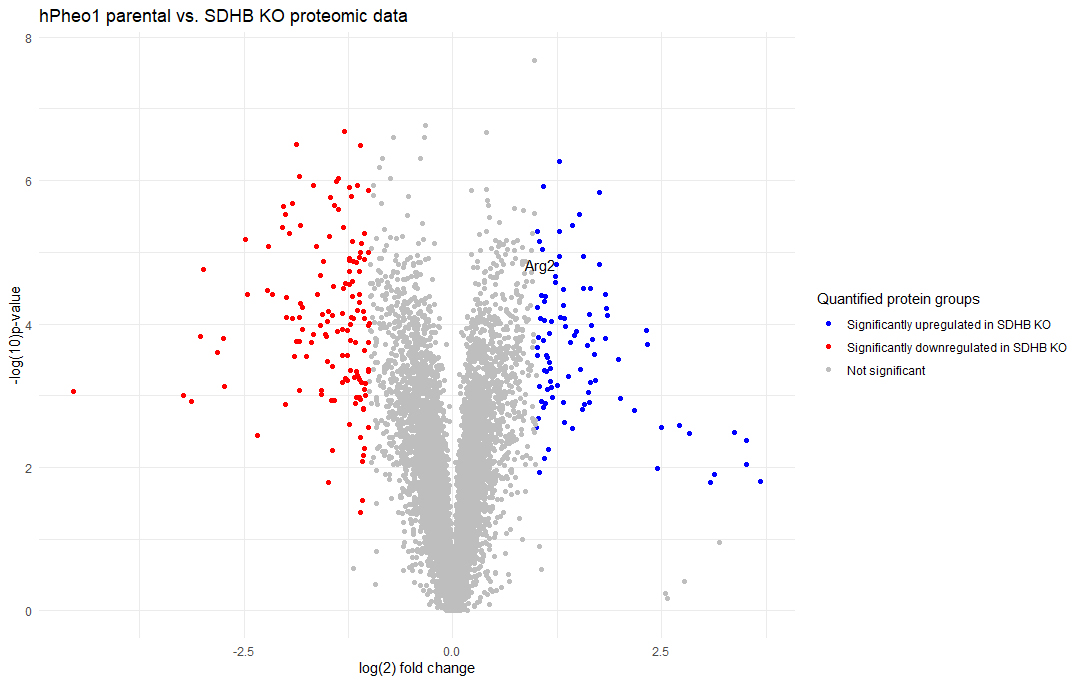
- Proteodata pro tvorbu volcano plotu
- Correlation pro srovnání a korelaci proteomického a transkriptomického datasetu
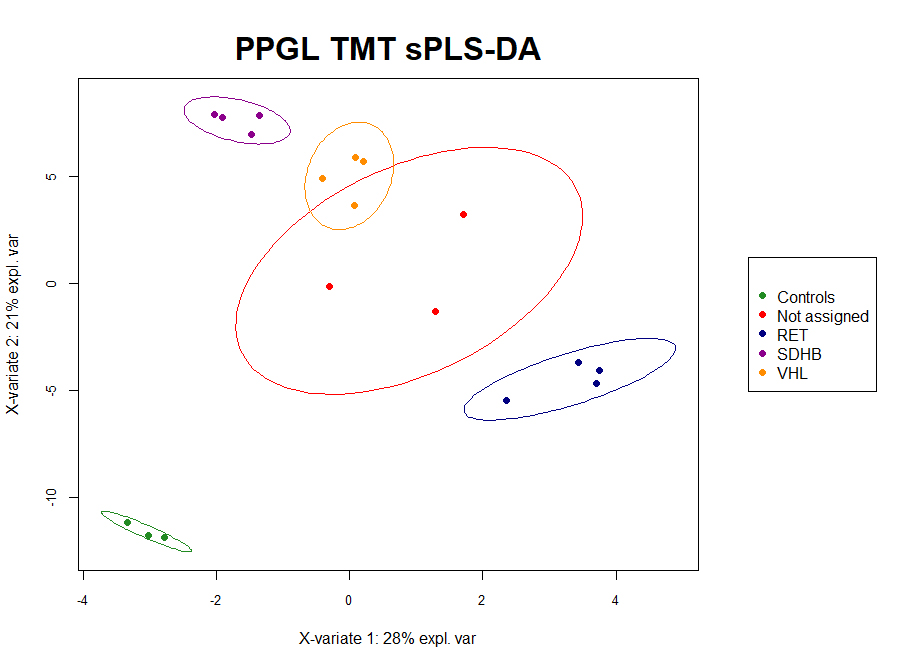
- sPLS-DA-PCA pro analýzu proteomických dat metodami sPLS-DA a PCA
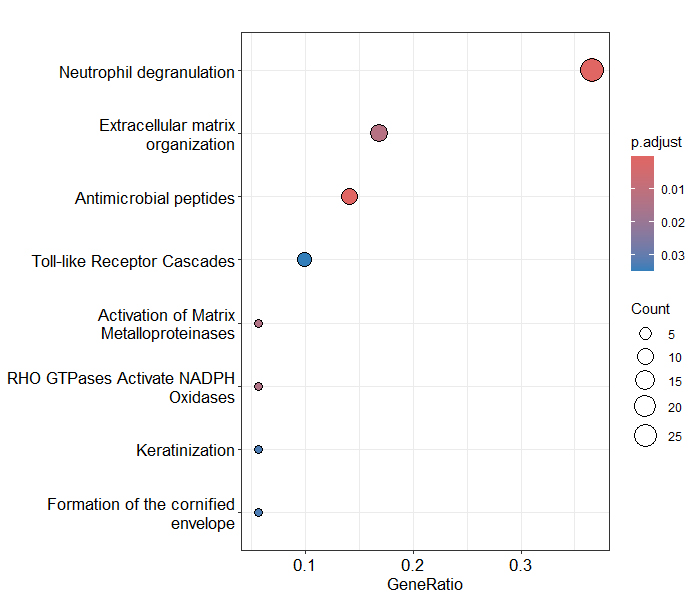
https://github.com/hwllffrdd/convert_enrich
Toto je zase taková skromná pipeline umožňující využití funkce enrichPathway na proteomická data.